El descubrimiento de los genes partidos
En 1993, el Premio Nobel de fisiología y medicina (hyperlink) se otorgó a Richard Roberts y Phillip Sharp por sus estudios acerca de los genes partidos y fragmentación del ARN, uno de los mecanismos fundamentales implicados en la diversidad de proteínas y la evolución.
En 1972, Richard Roberts inició su trabajo de investigación básica acerca de la regulación genética y estructura del Adenovirus (hyperlink) lo que llevó al descubrimiento de la fragmentación de genes en 1977. Resulta bastante notable el hecho de que Richard Roberts y Phillip Sharp, cada uno trabajando de forma independiente, al mismo tiempo, con el mismo modelo orgánico y las mismas técnicas dieron con la teoría de empalme genético: parte de la información genética almacenada en el ARN se corta y empalma antes de traducirse a proteínas.
En células eucariotas el ADN se transcribe a ARN mensajero (ARNm) en el interior del núcleo. Desde de ahí, el ARN se transporta de modo selectivo hacia el citoplasma dónde se traduce a proteínas. Esta compartimentalización del proceso de transcripción permite que los genes se procesen de un modo más exhaustivo que en procariotas.
Cuando Roberts y Sharp compararon el ARNm del núcleo y el del citoplasma, encontraron que eran distintos. Además, cuando compararon los segmentos de ARN citoplasmático con los segmentos de su ADN de procedencia mediante la creación de híbridos de ARN y ADN (hyperlink), hallaron que los segmentos de cada uno no eran copias perfectas de su segmento complementario. Algunas regiones sí correspondían mientras que otras simplemente parecían haber desaparecido en el ARNm del citoplasma.
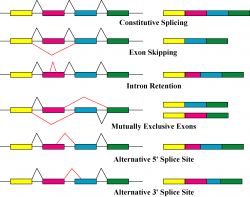
Estudios anteriores sugerían que durante el procesamiento del ARNm, algunas regiones nucleótidas se cortan y empalman. Son los llamados intrones (hyperlink). Otras regiones de ARNm se mantienen juntas formando las regiones que codifican la transcripción de proteínas. Esta parte del ARN mensajero se conoce como exon (hyperlink).
Algunos estudios usando mutaciones identificaron 'secuencias consensuadas' en el ARNm, secuencias de reconocimiento, puntos de corte y unión en el ARNm que limitan los intrones y los exones y que son las responsables del empalme genético. Las mismas secuencias se hallaron en mamíferos, levaduras y plantas. Este alto nivel de conservación data el proceso de traducción de proteínas a miles de millones de años coincidiendo con los orígenes de la célula eucariota.
El empalme puede producirse con o sin la ayuda de un empalmosoma (una gran y compleja maquinaria molecular) y en tripanosomas, C.elegans y vertebrados se han identificado distintos tipos de empalmes, cis o trans (hyperlink).
Los empalmes genéticos son habituales en genomas por dos buenas razones. En primer lugar, resulta realmente sencilla la aparición de nuevas combinaciones que pueden crear nuevos genes o proteínas mediante la duplicación de exones (hyperlink). Por ejemplo, un exon que contenga un dominio activo para una enzima puede duplicarse, moverse y asociarse con otra importante función de otro exon, creando así una proteína enteramente nueva, casi como si se construyeran bloques. En segundo lugar, de este modo un gen puede también codificar múltiples proteínas (hyperlink). Por ejemplo, si un gen contiene 8 exones, puede asimismo fabricar una proteína juntando los exones 1,4 y 6 o asociando exones 2,4,6 y 7. Cada combinación dará como resultado la creación de dos proteínas completamente distintas.
Esta es una de las explicaciones del porqué no existe una correlación directa entre la diversidad de los genes y la de las proteínas.
Last edited: 8 May 2015 14:49
